El Centro de Coordinación de Alertas y Emergencias Sanitarias (CCAES) del Ministerio de Sanidad está desarrollado un protocolo, en colaboración con las comunidades autónomas, para integrar la secuenciación genómica en el sistema de vigilancia de coronavirus SARS-CoV-2. Esta iniciativa surge ante la aparición de "algunas mutaciones o combinaciones de mutaciones [que] pueden proporcionar al virus una ventaja selectiva, como una mayor transmisibilidad a través de un aumento en la unión del receptor".
Según detalla el protocolo, Integración de la secuenciación genómica en la vigilancia del SARS-CoV-2, al que ha tenido acceso El Independiente en una versión del 13 de enero, se subraya también la "preocupación acerca de la aparición de una variante que escape al efecto de los anticuerpos neutralizantes generados tras una infección previa o tras la vacunación, lo que podría condicionar casos de reinfección o pérdida de la eficacia vacunal".
El documento repasa algunas de las variantes del coronavirus que más éxito en su dispersión han tenido, como la variante con la mutación D614G en la proteína S que se impuso en España, la variante británica que es la predominante en Inglaterra o la variante sudafricana.
El CCAES lo considera "un problema de salud pública de primer orden que puede repercutir de forma importante en el control de la pandemia"
El CCAES considera "un problema de salud pública de primer orden que puede repercutir de forma importante en el control de la pandemia" la aparición de variantes que aumenten la transmisibilidad del virus, su virulencia o que escapen a la acción de los anticuerpos neutralizantes generados tras la infección natural o la vacuna.
Siguiendo las recomendaciones del Centro Europeo de Enfermedades Infecciosas (ECDC, en sus siglas en inglés) Sanidad ha desarrollado un sistema de muestreo "debido a la premura originada por la alerta que supone la aparición de la variante VOC 202012/01 y sus implicaciones en salud pública y teniendo en cuenta la capacidad actual de los laboratorios y la todavía inmadurez del sistema centinela".
El sistema de muestreo coordinado por el Centro Nacional de Microbiología del Instituto de Sanidad Carlos III incluye muestras representativas de todas las comunidades autónomas. Cada comunidad designa un laboratorio centinela o dos, si es pluriprovincial, con capacidad de realizar la secuenciación o, en su defecto de enviarlo a un laboratorio.
La muestra se ha definido de manera semanal, cada miércoles se recogerán los primeros casos positivos de ese día de Covid de las comunidades autónomas. El 80% procederán de Atención Primaria y el 20% restante de hospitales. El número de casos secuenciados variará en función de la incidencia. Si la incidencia es baja, menor de 150 casos cada 100.000 habitante, habría que secuenciar 157 muestras en toda España (ver tabla). A día de hoy estamos en la máxima incidencia de este protocolo, por lo que se tendrían que secuenciarse 298 casos positivos para buscar las peligrosas nuevas variantes.
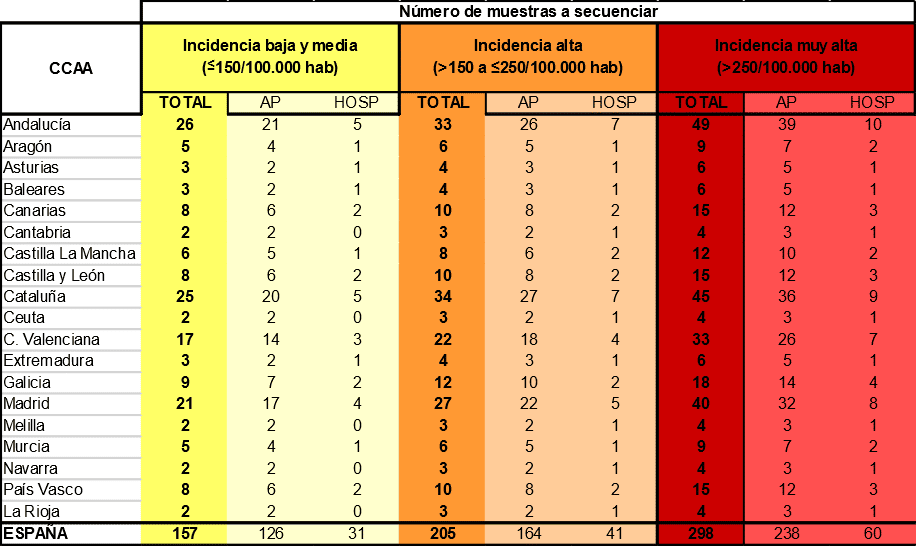
Los resultados de las comunidades se deben incluir en el SiViEs, el sistema de información del sistema nacional de salud. Según ha confirmado Sanidad a El Independiente se sigue trabajando para hacer los ajustes de manera que se puedan integrar los resultados de las secuenciaciones de las comunidades en la información de la pandemia. A día de hoy Sanidad tiene notificados 147 casos de la variante británica.
Con este cambio España introduce un sistema de vigilancia genómica que varía respecto al programa de SeqCOVID, un proyecto que ha situado a nuestro país como tercero de la Unión Europea y sexto del mundo en número aportaciones a GISAID la iniciativa global que hace seguimiento de la evolución del coronavirus. Pero la capacidad de mutación del virus requiere un sistema más ágil para poder detectar la expansión de una variantes que podría determinar la evolución de la tercera o hacer peligrar el trabajo de vacunación realizado.
Te puede interesar
Lo más visto